Team:Kyoto/projectRNA
From 2013.igem.org
(→Structure Prediction) |
(→Structure Prediction) |
||
| Line 175: | Line 175: | ||
===Structure Prediction=== | ===Structure Prediction=== | ||
[[File:2013IGKUprojectRNAfusionCENTROIDattenuatoraptamer.png]] | [[File:2013IGKUprojectRNAfusionCENTROIDattenuatoraptamer.png]] | ||
| - | [[File:antisense_spinach]] | + | [[File:antisense_spinach.png]] |
| - | + | <br> | |
===Conclusion=== | ===Conclusion=== | ||
Revision as of 23:21, 27 September 2013
count down
Contents |
RNA Oscillator
Introduction
Motivation
先のチューリングの例でみたとおり、場のスケールを多數の大腸菌で考えると菌体密度のようなfactorが影響してきてしまう。そのため、反応が複雑になってしまい、wetとdryの乖離を進める一因となっている。そこで、主要なファクターを考えやすくするために、場のスケールを一細胞で考えようと試みた。ここで扱うモデルは、よく研究されているオシレーションである。
As we state in Turing Model Project, if we try to make turing pattern with many cells of E.coli, we have to consider irregular factors such as density of cells. So, we have to simulate too complex circle to be practical. This complexity makes wet and dry unmatchable. So, then, we tried to compose turing pattern model on the inside of each E.coli’s cell. We adopted well-researched oscillation model.
細胞内のオシレーションとしては、例えばシアノバクテリアのkai protein familyなどがある<reference>。Kaiタンパクのオシレーションの機構は<ふにゃふにゃ>である。しかし、kaiのオシレーションを大腸菌で実現しようとすると。周期の時間がスケールが大腸菌の分裂速度よりも大幅に違う。よって、正確にモデリングすることが困難だろう。だから、短時間のモデルが考えやすいだろう。
In the natural world, for example, kai protein family oscillates in the cell of cyanobacteria. <reference> Kaiタンパクのオシレーションの機構は<ふにゃふにゃ>である。However, because the cycle of kai protein family’s oscillation is much longer than E.coli’s cell cycle, it is difficult to create precise model of kai protein in E.coli. Hence, quicker oscillation is easy to simulate.
さて、チューリングのモデルを大腸菌内で、かつ短い時間スケールで実現する際のfactorとして、私たちはRNAを提唱する。実際、転写調節因子としてのRNAの研究は始まっている<reference>。RNAを使う利点としては、次の2点が挙げられる。
Then, as quickly-oscillating factor in E.coli, we advocate functional RNA. In fact, research of RNA to regulate transcription have been undertaken. <reference> The merits of adopting functional RNA is following:
<引用>4RNAは二次構造の予測や、RNA同士やDNAに対する特異的な結合を可能にするような設計を行うこともタンパク質に比較すると容易である。よって、遺伝子回路を製作するにあたって、回路を構成するRNA同士が塩基配列特異的な相互作用をするように設計すれば、数に限りがある既存のアクチベーターやリプレッサータンパク質を用いては不可能だったような、一細胞内で複数の独立した回路を共存させるということが可能になる。加えて、回路に直接関係しない任意の遺伝子の発現量をそれ同調させることも可能となる。
Firstly, compared to protein, it is easier to predict the secondary structure of RNA, and to design the structure in order to bind to a specific RNA or DNA. Therefore, if we design the RNA which constructs the circuit to interact specifically to the base sequence, we can make some different circuits co-exist inside one cell. Moreover, since we can predict the structure we can link a post-transcriptional RNA reporter to the functional RNA to stop the conformational alternation, and realize an imaging of the RNA.
5さらにRNAは転写後、機能するまでに翻訳の時間を要しないため、応答までの時間が短縮される。また、生体内での分解もタンパク質と比較して早いので、転写調節から応答までの時間を比較的短くすることも可能になると考えられる。そのため、遺伝子回路を構成する分子を決定するとき、 タンパク質とRNAを適宜織り交ぜることで、転写調節から目的分子の細胞内の量を調節する時間をより広い幅でcontrolできるようになるかもしれない。</引用>
今回、私たちの用いるオシレーションのモデルは、<チューリングのアレ>です。このモデルにはactivatorとrepressorが必要です。それぞれの要素については以下に述べる。
Repressor
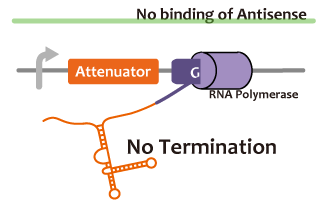
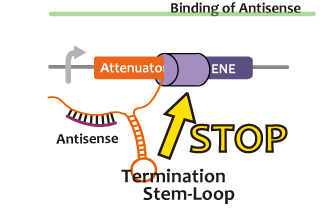
We took up non-coding RNA (ncRNA) complementarily binding mRNA as an example of functional RNA which repress transcription. Some kinds of ncRNA work as a transcriptional repressor in vivo, for example, Gram-negative bacteria Staphylococcus aureus regulates a copy number of plasmid called pT181 in this mechanism.5The ncRNA in pT181 plasmid controls the fate of transcriptional elongation in response to an input by antisense RNA. Attenuator region, which lies in 5' untranslated region of a transcript, folds into two different RNA structures. By an interaction with complementary antisense RNA, attenuator region forms Rho-independent terminator and the transcription of the downstream is stopped. Without antisense RNA, RNA in attenuator region folds into an alternative structure which allows transcription of the downstream (Novick, 1989). The uniqueness of this mechanism is that it is constructed with only RNA and without other small molecules, many synthetic biologist constructed a variant of it by means of nucleotide substitution etc. (Takahashi et al, 2013). We chose this mechanism in gene repression.
To ensure the function of antisense RNA and attenuator region, we will compare the amount of mRNA of GFP located in the downstream of an attenuator region in the presence and absence of antisense RNA.
転写の抑制を行うようなRNAの例として、我々は、伝令RNAに相補的に結合するncRNAによる転写制御を挙げる。これは、生体内でのRNAによるゲノム転写機構のひとつ、Gram-negative bacteria Staphylococcus aureusのpT181と呼ばれるplasmidなどのコピー数のregulationの機構である。RepressorとなるRNA (Antisense RNA)がある状態では、プロモーター下流のAttenuator locusがRho-independent terminator を形成することによりgenome coding部位の転写が抑制されるが、if the antisense RNA fails to bind, nascent RNA refolds into an alternative structure which prevents termination and promotes read-through (Novick, 1989) という仕組みを用いている。この機構は、他のリボスイッチと違いRNAのみで他の低分子化合物を用いていないため、合成生物学の新たな手法として、塩基置換などにより様々なタイプのものが作られている (Takahashi et al, 2013)。 われわれはこれをRepressionの回路とした。AttenuatorとAntisenseの、Attenuator Regionより下流の遺伝子の転写を阻害する機能を確認するため、Attenuator antisense RNAの存在下と非存在下で、Attenuator Region下流のGFP遺伝子の発現量を比較した。
In order to check the function of Attenuator and Antisense, we introduced Attenuator and Antisense into E.coli as experimental groups. We compared this E.coli with several controlled group in expression of GFP.
------const------
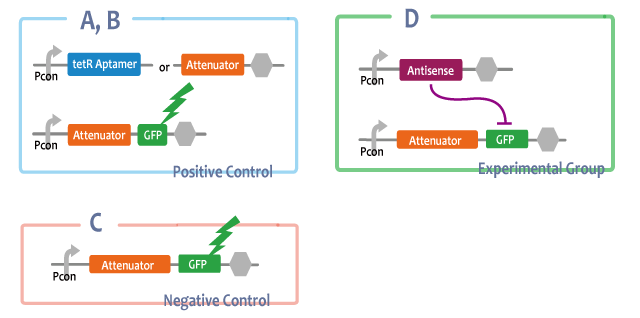
Positive Control
A-B: Pcon-RNAs(tetRaptamer, attenuator), Pcon-atte-GFP
-antisenseを他のRNAで置き換えたもの。これによってRNAであることが問題なのでなく、antisenseのもつ相補的配列が問題であることを確かめる。
C: Pcon-atte-GFP
Antisense非存在下においてはGFPの発現は抑制されないことを確認する。
Experimental Group
D: Pcon-antisense Pcon-atte-GFP
Positive Control
A-B. Pcon-RNAs(tetRaptamer, attenuator), Pcon-attenuator-GFP
In order to check the uniqueness of Antisense in repression, we introduced other RNAs into E.coli.
C. Pcon-attenuator-GFP
This E.coli shows that if there is no Antisense, the expression of GFP is not repressed.
Experimental Group
D. Pcon-antisense Pcon-attenuator-GFP
------const------
---figcaption----
Antisenseが常時発現している大腸菌(figE)においてはAttenuator Regionの下流にあるGFPの転写が抑制され、Antisenseが存在しない大腸菌(figD)では抑制されていないことから、figEの大腸菌における転写抑制はAntisenseに起因することがわかる。figEの大腸菌でAntisenseをコードしていた部分を他の配列に置き換えた大腸菌(figA-C)におけるGFPの転写量はAntisenseを転写しない大腸菌(figD)に比べて遜色ないことから、figEの大腸菌での転写抑制はAtternatorに特異的なものであったことが導かれる。
Compared with E.coli which didn’t express Antisense(figC), E.coli which always expresses Antisense(figD) was repressed in expression of GFP. This demonstrates that this repression was caused by expression of Antisense. Furthermore, because E.coli in which other structure of RNA was introduced(figA-B) expresses as much GFP as E.coli which didn’t expresses Antisense, we can say that this repression was peculiar to Antisense.
---figcaption----
Activator
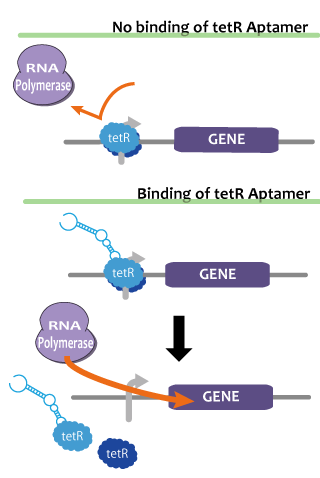
We pick up tetR aptamer as an example of functional RNA which activates transcription. TetR aptamer specifically binds tet represser (tetR), which binds DNA specific site, repress transcription of downstream gene, and induce tetR conformational change and tetR reorientation.7 That is, if in one cell, tetR is constantly expressed, gene located in the downstream of tet promoter is usually repressed and only when tetR aptamer is being expressed, it derepressed and transcribed.
In our experiment, we check and measure tetR aptamer’s function in E.coli by comparing GFP fluorescence regulated by tetR promoter and GFP expression level by qRT-PCR in the following 4 cellular cases:1 TetR and tetR aptamer is constantly expressed. 2 Only tetR is constantly expressed and tetR aptamer is not induced.3 TetR and other functional RNA is constantly expressed. 4 TetR is not induced.
転写のアクチベーションを行うような機能性RNAの例として、我々はtetR aptamerを挙げる。これはtet repressorに特異的に結合するアプタマーであるが、DNAの特定領域に結合して転写を抑制しているtet repressorに結合してDNAから解離させる作用も持つ。つまり、常に一定量のtet repressorが発現し、存在しているような細胞内では、tetR aptamerが発現している間のみtet promotor以下の転写の抑制が解除、つまり活性化され、tetR aptamerが発現していず存在していない場合は、tetRの機能によって転写が抑制されるようになる。 tetR aptamerの働きを確認するため、tetRタンパク質とtetR aptamerを常時発現させた場合と、tetRタンパク質のみを常時発現させた場合、tetRとtetR アプタマー以外の構造をもつRNAを発現させた場合、tetRを発現させなかった場合とで、tetプロモーター下流に配置したGFP遺伝子を発現させその蛍光を見、qRT-PCRで発現量を比較しtetR aptamerの働きを確認した。(顕微鏡で蛍光度の差が確認できたときはqRT-PCRは補強扱いとし、確認できなかった場合はqRT-PCRのみを蛍光度の比較の尺度とする。)
In order to check the function of tetRaptamer, we introduced tetR and tetRaptamer as experimental group. We compared this E.coli with several controlled groups in expression of GFP.
-----コンストラクション------
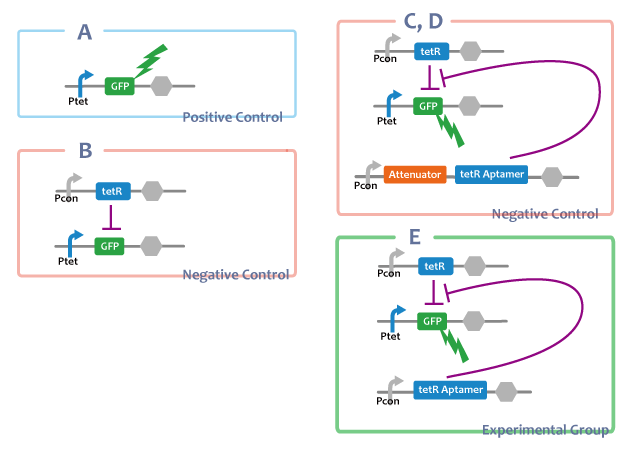
Positive Control
A. Ptet-GFP
-tetRを導入せず、Ptet-GFP単体のもの。tetRが存在しない場合にPtetがonになるということの確認。
Negative Control
B. Ptet-GFP, Pcon-TetR
-tetRaptamerが存在しない場合。tetRがそのままで転写抑制をすることの確認。
C-D. Ptet-GFP, Pcon-TetR, Pcon-RNAs(anti_attenuator, attenuator)
-tetR aptamerを他のRNAで置き換えたもの。これによってRNAであることが問題なのでなく、tetR aptamerのみが持つ構造と機能が問題であることを確かめる。
Experimental Group
E. Ptet-GFP, Pcon-TetR, Pcon-tetRaptamer
Positive Control
A. Ptet-GFP
In order to check whether the GFP gene is expressed when there is no tetR protein, we introduced only Ptet-GFP.
Negative Control
B. Ptet-GFP, Pcon-tetR
Through this E.coli, it is confirmed that when tetR is expressed, tetR surpresses the expression of GFP.
C-D. Ptet-GFP, Pcon-tetR, Pcon-RNAs(antisense, attenuator)
These kinds of E.coli shows that the surpression of the function of tetR is caused only by tetRaptamer.
Experimental Group
E. Ptet-GFP, Pcon-tetR, Pcon-tetRaptamer
-----コンストラクション------
-----fig Caption------
tetRを発現しない大腸菌(figA)の蛍光はtetRを発現する大腸菌(figB)のそれよりも強いことから、tetRはtetリプレッサーに結合して下流の転写を妨げることがわかる。tetRを発現し、tetRaptamerを転写しない大腸菌(figB)のGFP転写量に比べてtetRとtetRaptamerの両方を発現する大腸菌(figF)のGFP転写量が有意に大きいことから、tetRaptamerはtetRによる転写の抑制を解除する働きがあることが示唆される。tetRaptamerをコードしていた部分を他の配列に置き換えた大腸菌(figC-E)の蛍光はtetRとtetRaptamerの両方を発現する大腸菌(figF)よりも弱く、tetRのみを発現する大腸菌(figB)と同程度であることから、figFの大腸菌におけるtetRの機能の抑制はtetRaptamerに特有のものであることがわかる。
The fact that the fluorescence of E.coli which expressed tetR(figB) was weaker than E.coli which didn’t express tetR(figA) shows that tetR represses the expression of genes at the downstream of tet promotor. Since E.coli introduced tetR and tetRaptamer (figF) expressed more GFP than E.coli introduced only tetR(figB), we were confirmed that tetRaptamer cancels the effect of tetR {in some degree / almost completely}. Moreover, E.coli which was introduced other structure of RNA(figC-E) could express as much GFP as E.coli introduced tetR only(figB), which demonstrates that the surpression of the function of tetR protein is unique to tetRaptamer.
-----fig Caption------
Reporter
我々は、RNAでできたレポーターとなりうる分子として、Spinachを挙げる。これはJeremy S. Paige, Karen Y. Wu, Samie R. Jaffrey, によって設計されたアプタマーの一種で、GFPを模倣している。SpinachはGFPの蛍光部位によく似た合成物であるDMHBIに特異的に結合するアプタマーから設計された。GFPのfluorophoreはdenatured GFPでは蛍光を示すことがなく、分子の奥に折りたたまれて初めて蛍光を発するようになる。DMHBIもこれと似た性質を持っており、単体ではほぼ蛍光を示すことはなく、GFPの構造の持つ機能を真似たSpinachの高次構造の奥に取り込まれて初めて蛍光するようになる。そのため、サンプルにDMHBIを加えた後に蛍光を確認すると、サンプル内にSpinachが存在するかどうかがわかる。もし存在すればSpinachはDMHBIと結合して蛍光を発するだろうし、存在しなければ蛍光は発しえない。Spinachを用いることで、RNAを直接イメージングできる他、安定なタンパク質では確認できない、大きく変化するRNAの発現量を正確に反映することが出来る。
Spinach is an example of a reporter molecular, which is a kind of aptamer designed by Jeremy S. Paige, Karen Y. Wu and Samie R. Jaffrey imitating GFP. It is designed from aptamer combining with the complex--DMHBI which is considerably similar to the fluorescence site of GFP. The fluorescence of GFP show as long as the molecular is folded in the back of the molecule, instead in denatured GFP. DMHBI also shows almost the same property--the simple substance produces little fluorescence, compared with when captured by higher-order constructions. That is to say, if fluorescence is conformed after DMHBI is added, it is manifest that Spinach exists. If Spinach exists, it combines with DMHBI to produce fluorescence, vice versa. Hence, by using Spinach, it’s possible not only to image RNA directly, but also accurately reflect the expression level which vary intensely, which can’t be confirmed via stable protein.
We suggest Spinach as a molecule which can be a reporter made of RNA. It is an aptamer designed by Jeremy S. Paige, Karen Y. Wu, Samie R. Jaffrey and imitates GFP. Spinach was designed from the aptamer which binds specifically to DMHBI, which is similar to fluorophore of GFP.
【メモ:Assay、Result、Discussion】
Fusion
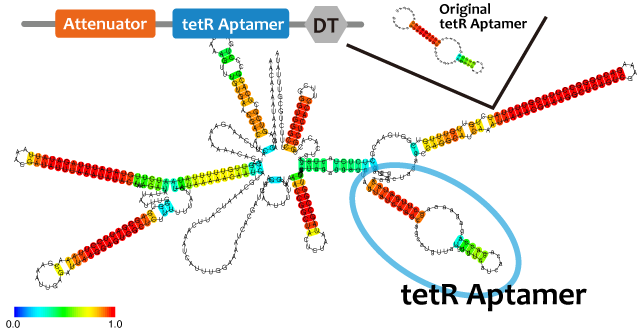
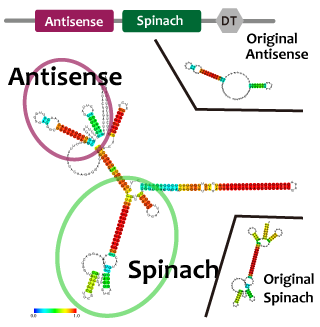
これらを使って遺伝子回路を組み立てるとき、複数のmoduleを同じ機能要素に組みこまなければならないときも十分あり得る。例えば転写抑制の様子をレポートするとき、異なる因子で促進と抑制を行うような系を作るときである。このとき、複数のモジュールを連結したことによる相互作用や立体構造の問題により機能が阻害される可能性がある。タンパク質ではその問題を予測するのは難しいが、RNAであれば配列情報から比較的簡単に二次構造を予測することができ、これらの問題を回避出来る。われわれは、機能を確認したtetR aptamer, Antisense-Attenuator RNA, をそれぞれつなぎあわせ、二次構造を予測し、実際に働いていることを確認した。tetRタンパク質存在下でtetR aptamerとAttenuator antisense RNAを組み合わせたRNAがPtetプロモーター下流のGFPの転写量を増加させるかを確認した。
並びにAttenuator antisense RNAとSpinarchを連結したRNAを発現させ、Attenuator Region下流のGFP遺伝子の発現量が減少していることとSpinarchがDFHBI存在下で蛍光するかどうかを確認した。
Intending to check the process of transcriptional repression system and the system which promotes and represses processes of transcription by using different factors, we have to join some modules into a single RNA strand.
When we combine plural modules, the function of the modules may be inhibited by interactions and steric structures between each other. in the case of RNA,it is easier to predict and avoid the steric problems than that of proteins because we can predict the secondary structure of RNA from its primary structure. We combined tetR aptamer and Antisense-Attenuator RNA, whose functions are confirmed, and predicted secondary structures, as a result it actually worked. We also observed tetR aptamer-Attenuator antisense fusion RNA increased expression level of downstream GFP of tet promoter in the presence of tetR proteins.
-----const-------
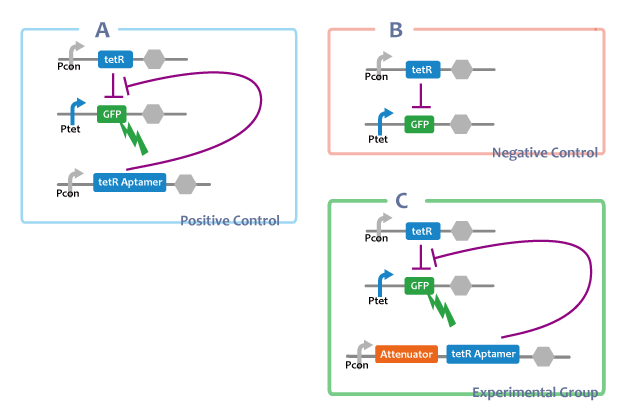
experimental group
a. Pcon-atte-tetRaptamer-DT Ptet-GFP-DT Pcon-tetR-DT
positive control
b. Pcon-tetRaptamer-DT Ptet-GFP-DT Pcon-tetR-DT
-Fusionする前とのtetRaptamerの働きの比較
negative control
c. Ptet-GFP-DT Pcon-tetR-DT
Positive Control
A. Pcon-tetRaptamer Ptet-GFP Pcon-tetR
Through this E.coli, we can confirm that separated tetRaptamer restricts the function of tetR protein.
Negative Control
B. Ptet-GFP Pcon-tetR
This E.coli shows that tetR protein represses the expression of genes at the downstream of tet promotor.
Experimental Group
C. Pcon-atte-tetRaptamer Ptet-GFP Pcon-tetR
------const-------
We used centroid fold (URL) and mfold (URL) to predict the secondary structure of RNA(a). As the picture shown below, the structure of tetR aptamer is not affected by attenuator stem loop. This suggests that the efficiency of tetR aptamer is not affected by the existence of attenuator stem loop.
Centroid fold, mfoldのfig-tetR aptamer only----tetR aptamer-antisense
tetRが常時発現されている状態では、AttenuatorとtetRaptamerを連結したRNAを転写する大腸菌(figC)のGFP発現量は、tetRaptamerを転写しない大腸菌(figB)よりも多く、ほかのRNAと連結していないtetRaptamerを転写する大腸菌(figA)と比較して{ほぼ同等 or 小さい}であることから、AttenuatorとtetRaptamerを連結すると、tetRaptamerは{全く干渉せずに機能する or 効果は下がるが機能する}ことがわかる。
If tetR is expressed, E.coli in which the united RNA(figC) was introduced expresses more GFP than E.coli which didn’t have the tetRaptamer sequence(figB). By comparing E.coli expressing independent tetRaptamer(figA) and E.coli expressing the united RNA(figC), it is convinced that tetRaptamer next door to Attenuator { works as well as independent one / works more weakly than a independent one. However, it certainly works.}
Experiment
(・Construction) ・転写確認 ・構造予測
Result
(・コンスト泳動結果)
RT-PCR
RT-PCR
File:ElectrophoresisRT
Structure Prediction
Conclusion
--RTでコンストの確認出来た
--RT-PCRでの転写(機能)確認できた
--Fusionの二次構造見た 影響しなさそうだった
wetで事実作れる準備ができた
future work
・定数探し ・シュミレーションする ・Attenuator-tetR アプタマーのコンストラクション ・機能確認 ・実際にwetでオシレーションができることをみる ・wetとdryを比較する
文章のたまり場
旧conclusion
In this project we confirmed the function of activator (tetR / tetR aptamer) and repressor (Attenuator region / Antisense). Moreover, we predicted the secondary structure of linked RNA (Antisense RNA-tetR aptamer) to check the influence of linkage to the structure, and finally we confirmed that actually the function of tetR aptamer do not lost. Our outcome of this project will directly connects to the progress of synthetic biology, especially constructing gene circuits. These two types of functional RNA will play important role when we regulate gene expression in peculiar gene cycle. For example, we may regulate a gene circuit which contains rapid gene transcriptional cascades by these RNA modules.
Future Work
To show this possibility, we designed a gene circuit which uses these RNA module. This circuit produces transcriptional oscillation. Oscillation circuits are important and essential gene circuits in many organisms and always be in the center of synthetic biology, therefore it is suitable for the cutting edge of new type of gene regulation.
When it comes to oscillation, we have to have a module which acts as reporter to show the changing amount of post-transcriptional RNA. Usually, protein reporters such as GFP are used for this purpose. However, in this circuit protein reporters may not be able to be used, because of the length of the period of the oscillation. Because RNA’s degradation is so fast and RNA do not need to be translated or folded like protein, the period of oscillation should be too short. According to XX who constructed similar gene circuit using RNA modules, this kinds of circuit produces 10 minutes cycle reaction. This means protein degradation is too slow (takes XX hours even with the degradation tag) [要出典] to image this RNA oscillation.
To solve this problem, we will suggest a new RNA module, which called spinach. This is a kind of aptamer, which is designed by Jeremy S. Paige, Karen Y. Wu, and Samie R. Jaffrey.20 They imitated the structure of GFP in this project. The designing of Spinach is changing the structure of an aptamer which specifically combines with DFHBI, which has similar structure to fluorophore of GFP. Denatured GFP doesn’t have fluorescence. Only if GFP is folded correctly, the fluorophore of GFP, which is in inner area, emits fluorescence. Therefore, we can confirm whether there is Spinarch in a sample by adding DFHBI. If the sample contains Spinarch, the sample will emit fluorescent. Vice versa. Spinach may degrade first enough for the oscillation, therefore we propose this for reporter of this oscillation.
The circuit of oscillation which uses spinach and the two RNA module is like below. This describes mechanism of producing oscillation.
この回路がオシレーションを形成する仕組みは、以下のようになっている。初期条件として、Constitutive Promoterにより合成されたTetRにより、Ptetはrepressされている。 オシレーションの開始はPtet下流のPlacがIPTGにより誘導されることである。これによってRNA-Actが合成開始され、その中のtetR aptamer配列がPtetをactivateする。 ActivateされたPtetはさらにRNA-Actを合成し、ここでポジティブ・フィードバックがかかることでRNA-Act, RNA-Repともにその量を増やす。すると、RNA-Repの配列内のSpinachにより緑色蛍光が確認される。 RNA-Repの量が十分に増えると、そのAttenuator antisenseの部位がRNA-ActのAttenuator locusに結合し、RNA-Actの転写量を減少させる。 するとTetR-AptamerによるActivationが小さくなることで、RNA-Act, RNA-Repの量が減少する。すると、Spinachによる蛍光は減衰する。 RNA-Repの量が十分に減少すると、Attenuator antisenseによる転写抑制が解かれ、再びRNA-Actの転写量が増えることとなる。これが繰り返されることで、オシレーションを作り上げている。この回路からは、RNAならではの分解・生成が速い性質によって、10分周期程度の短いSpinach蛍光のオシレーションを生むことが出来ると予測できる。
This circuit oscillates in the following way: First, tet promoter is repressed by tetR at the downstream of constitutive promotor. Then, the oscillator is turned on by IPTG. IPTG activates Plac and tetRaptamer, Spinarch, and Antisense at the downstream of Ptet which are transcribed. Because tetRaptamer activates tet promotor, positive feedback occurs and more and more tetRaptamer, Spinarch, and Antisense are accumulated. Then, this circuit gets fluorescence. After Antisense is accumulated to some extent, tetRaptamer, at the downstream of Atteruator region, is repressed. Then, because new tetRaptamer is not created, the amount of tetRaptamer decreases quickly. So, tet promotor is repressed by tetR protein and the amount of Antisense and Spinarch falls, too. Then, this circuit loses fluorescence. After the amount of Antisense decreases sufficiently, this circuit recovers first condition. Through this cycle, this circuit acts as an oscillator. Since RNA is generated and resolved quickly, this circuit should oscillate as quick as about in 10 minutes' cycle.
☝☝たぶん時制がめちゃくちゃですごめんなさい(´._.`)
Achievement
我々は、このプロジェクトで以下のことを達成した。
①
②
③
④
⑤
⑥
Parts List
<groupparts>iGEM013 Kyoto</groupparts>
Reference
[7(仮)][http://www.ncbi.nlm.nih.gov/pubmed/19246008 Anke Hunsicker et al.(2009)"An RNA aptamer that induces transcription"Chem Biol,16(2),173-180]
[20(仮)][http://www.sciencemag.org/content/333/6042/642.abstract Jeremy S. Paige et al.(2011)"RNA Mimics of Green Fluorescent Protein"Science Vol. 333 no. 6042 pp. 642-646]
[5(仮)][http://www.ncbi.nlm.nih.gov/pubmed/23761434 Melissa K. Takahashi and Julius B. Lucks.(2013)"A modular strategy for engineering orthogonal chimeric RNA transcription regulators"Nucleic Acids Research 41(15),7577-88]
 "
"